ESMFold è un modello di intelligenza artificiale sviluppato da Meta che, seppur usando metodi diversi, raggiunge risultati comparabili a quelli di Alphafold. Alphafold di DeepMind/Google è solo l’ultimo di una serie di sistemi di intelligenza artificiale che, dopo aver guadagnato un enorme successo, si sono visti insidiare il primato da concorrenti altrettanto performanti e agguerriti. Il sistema in grado di prevedere il ripiegamento proteico è stato recentemente sfidato da GPT-3 e DALL-E di OpenAI/Microsoft sono altri due famosi modelli che hanno subito una concorrenza simile, sia da parte di Google sia da Meta. In questi ultimi anni le multinazionali “big tech”, in particolare Google, Meta e Microsoft, si stanno sfidando a colpi di AI proprio in quei campi dove una delle tre sembrava aver stabilito lo stato dell’arte.

Indice degli argomenti:
I modelli per prevedere il ripiegamento proteico
Alphafold è un sistema di intelligenza artificiale in grado di capire la struttura tridimensionale delle proteine, sviluppato pochi anni fa da un team di ricercatori della società di intelligenza artificiale DeepMind. Il sistema, poi migliorato nelle successive versioni, è in grado di analizzare le sequenze di amminoacidi e prevedere con precisione la struttura tridimensionale delle proteine. Una capacità, questa, che aiuterà la scienza a comprendere meglio le funzioni delle proteine e sviluppare nuovi farmaci per il trattamento di malattie.
Il problema non è banale. Conoscere la struttura di una proteina significa capire cosa fa. Una proteina è composta da una serie di amminoacidi che si ripiegano su se stessi in maniera complessa e in numerosi modi diversi. Vedere in che modo si sono ripiegati gli amminoacidi vuol dire avere sotto controllo il funzionamento della proteina, comprendendo ad esempio come si comporterebbe se venisse a contatto con un dato farmaco. Il ripiegamento proteico è in altre parole il cuore della biochimica. Per fare un esempio concreto e decisamente attuale, i vaccini contro la Covid-19 si basano proprio sulla proteina Spike che è presente sulla superficie del virus SARS-CoV-2 dandogli quell’effetto “corona”.
È proprio scoprendo la struttura della proteina che si è stati in grado di realizzare i vaccini. Ma non solo, si pensa che un errato ripiegamento proteico sia alla base di svariate patologie come il morbo di Alzheimer e il diabete mellito di tipo 2, il morbo di Parkinson o la fibrosi cistica. Conoscere la struttura dei cristalli proteici potrebbe essere la chiave per creare delle cure mirate.
Quando DeepMind ha dimostrato che Alphafold era in grado di risolvere questo annoso problema della biochimica, il mondo ha iniziato a guardare con estremo interesse le potenzialità che l’intelligenza artificiale avrebbe apportato alla ricerca medica, ma non solo. Tuttavia, a guardare non erano solo gli scienziati ma anche gli altri colossi della tecnologia, e non tutti erano contenti di restare in tribuna mentre Google (DeepMind è di sua proprietà) veniva acclamata dalla comunità scientifica. Non stupisce, quindi, la notizia che Meta poche settimane fa abbia rilasciato un diretto concorrente di Alphafold chiamato ESMFold – ESM sta per Evolutionary Scale Modeling – che secondo i suoi creatori è in grado di ottenere risultati comparabili a quelli di Alphafold.
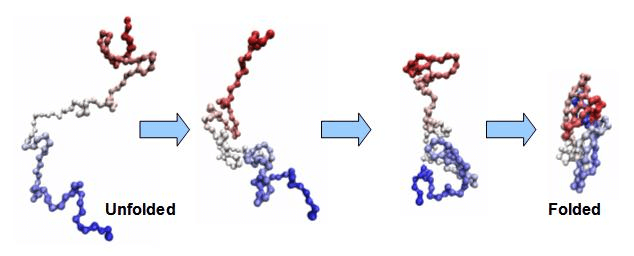
Proteine non ripiegate e ripiegate
EMSFold di Meta
Il team di Meta ha utilizzato metodi normalmente presenti nei modelli linguistici – come GPT-3, LaMDA e BLOOM – per addestrare il sistema di previsione su una scala più ampia di quanto sia stato fatto finora. I risultati hanno dimostrato che questi modelli linguistici erano in grado di prevedere con precisione la struttura tridimensionale di una proteina alla risoluzione dei singoli atomi. Il team di ricerca di Meta ha anche scoperto che, quando i modelli vengono scalati, essi apprendono informazioni aggiuntive che consentono loro di fare previsioni migliori.
Questa ricerca ha segnato un altro importante punto a favore dei modelli linguistici, che oggi sono prevalentemente usati per generare testo, intrattenere conversazioni ed estrarre informazioni da contenuti scritti. La generazione del testo, in particolare, guarda a cosa è stato scritto prima; quindi, suggerisce i termini che in base al suo addestramento avrebbero maggiore probabilità di seguire ciò che è stato scritto. Questa semplice caratteristica è stata applicata alle sequenze di proteine, consentendo al modello, precedentemente addestrato su una grande quantità di esempi, di prevedere come si dovrebbe evolvere la sequenza.
Come spiegano i ricercatori nel loro studio, l’ipotesi distributiva è una proposta della linguistica che suggerisce che il significato può essere dedotto dagli schemi delle parole in un testo. Questa idea è stata applicata alla deduzione di informazioni dalle sequenze in biologia, dove si propone che la struttura e la funzione di una proteina vincolino le mutazioni della sua sequenza, che sono selezionate attraverso l’evoluzione. Il fine ultimo è scoprire informazioni sufficienti dai dati di sequenza per modellare la complessità e la diversità delle strutture e delle funzioni biologiche.
Grazie a questo sistema, ESMFold riesce a uguagliare l’accuratezza di Alphafold, in particolare per le sequenze a bassa perplessità, ovvero più facili da prevedere, ma in maniera più rapida. Come spiegano sempre i ricercatori, poiché l’architettura di ESMFold consente al modello di essere eseguito interamente sulla GPU senza accedere a database esterni, ESMFold effettua una previsione su una proteina con 384 residui in 14,2 secondi, 6 volte più velocemente di un singolo modello AlphaFold2. Addirittura su sequenze più brevi il miglioramento è di circa 60 volte.

Concorrenza a colpi di modelli AI
L’esempio di ESMFold mostra come Meta abbia deciso una marcatura stretta per DeepMind, riaprendo una partita dove sembrava che l’azienda di Google avesse già stravinto. Ma si tratta solo dell’ultimo esempio di come le aziende BigTech, in particolare Google, Meta e Microsoft, abbiano deciso di sfidarsi a colpi di modelli AI, andando a creare insidiosi concorrenti per prodotti nei quali una delle tre aziende sembrava aver già chiuso la partita.
È il caso, ad esempio, dei modelli linguistici. Tutti conosciamo GPT-3, rilasciato nel 2020 da OpenAI (azienda in cui Microsoft ha investito pesantemente) e basato su precedenti iterazioni del modello. Per molti si tratta del modello linguistico de facto, il più discusso e il più usato. Ma piuttosto che lasciare la vittoria a Microsoft, in un campo peraltro dove i modelli promettono evoluzioni e scoperte eccezionali, Google ha deciso di sviluppare un modello ancora più performante chiamato LaMDA (quello che aveva indotto un tecnico dell’azienda a credere che fosse senziente), mentre Meta ha rilasciato il suo OPT-175, curiosamente delle stesse dimensioni di GPT-3, rendendolo open source.
Oppure possiamo citare DALL-E, sempre di OpenAI/Microsoft, un sistema di intelligenza artificiale generativa che consente di creare immagini partendo da una semplice riga di testo. Google ha subito rilanciato con Imagen e Meta si è aggiunta con il suo Make-a-scene, a riaffermare il concetto che nessuna delle aziende vuole lasciare campo libero ai concorrenti.
I modelli per il ripiegamento proteico sono dunque solo l’ultimo esempio di come queste tre aziende, ognuna in grado di condurre una eccellente ricerca di base in ambito intelligenza artificiale, non abbiano nessuna intenzione di consegnare una facile vittoria ai propri concorrenti.
Immagini generate da DALL-E
Conclusioni
Naturalmente non sono solo i grandi concorrenti multinazionali a preparare sistemi alternativi a quelli dell’incumbent. Alphafold poteva già vantare la presenza un sistema sfidante, noto come RoseTTAFold e sviluppato presso la Washington University, mentre GPT-3 ha già avuto numerosi concorrenti, fra cui GPT-NeoX-20B e il recente BLOOM LM, solo per citarne due.
In questi casi, tuttavia, il lavoro era stato svolto per così dire “dal basso”, ovvero da gruppi di ricerca senza grandi fini speculativi – in molti casi si è puntato all’open source – e decisamente senza il supporto vendite e marketing di cui può godere una multinazionale dai fatturati enormi. Concorrenti quindi con le armi spuntate, senza vere possibilità di imporre i loro modelli sopra quelli delle aziende big tech, che pertanto non impensierivano Microsoft e soci. La vera concorrenza, invece, sta arrivando dai rivali storici, e gli ultimi sviluppi ci hanno fatto comprendere che nessun ambito di applicazione AI è considerato off limits.






