Indice degli argomenti:
L’Ai spiegata agli umani
Quando si vuole rappresentare l’aiuto che l‘intelligenza artificiale potrà dare al benessere dell’umanità, uno degli argomenti più evidenti e indiscutibili riguarda la possibilità di scoprire nuovi farmaci. In un mondo sempre più digitale e robotizzato è per certi versi suggestivo che gli algoritmi e i modelli artificiali vengano adoperati per risolvere i problemi tipici della nostra natura biologica. In questo contesto la ricerca di DeepMind, con il rilascio del sistema di intelligenza artificiale AlphaFold, si pone esattamente al centro della questione.
DeepMind, cos’è e quali sono i suoi scopi
DeepMind è un’azienda creata a Londra dieci anni fa per sviluppare sistemi AI innovativi che in poco tempo entrò nei radar di importanti investitori. Quando Google la acquisì nel 2014, per 500 milioni di dollari, lo fece battendo Facebook, anch’essa interessata all’azienda creata da Demis Hassabis, Shane Legg e Mustafa Suleyman. Due anni dopo, nel 2016, DeepMind balzò agli onori delle cronache per AlphaGo, il sistema AI che batté il campione indiscusso di Go – Lee Sedol – in una sfida guardata da oltre 200 milioni di persone in tutto il mondo.
Già durante quella sfida Demis Hassabis, CEO di DeepMind, pensò che la tecnologia che avevano creato avrebbe potuto essere usata anche per risolvere uno dei più spinosi problemi della biologia: prevedere il ripiegamento proteico partendo dalla sequenza di amminoacidi.
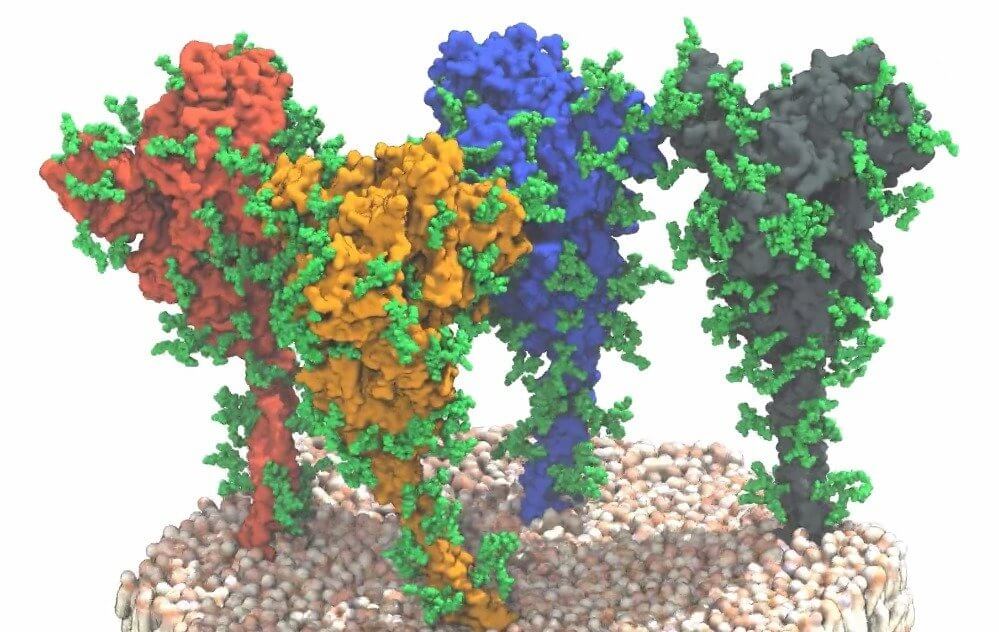
Proteine S (Spike) del SARS-CoV-2. © MPI f. Biophysik/ von Bülow, Sikora, Hummer
Cosa fa una proteina
Conoscere la struttura di una proteina significa capire cosa fa. Una proteina è composta da una serie di amminoacidi che si ripiegano su se stessi in maniera complessa e in numerosi modi diversi. Immaginiamo una struttura enormemente complessa, dove una piega in una direzione anziché in un’altra implica un comportamento completamente diverso della proteina. Vedere in che modo si sono ripiegati gli amminoacidi vuol dire avere sotto controllo il funzionamento della proteina, comprendendo ad esempio come si comporterebbe se venisse a contatto con un dato farmaco. Il ripiegamento proteico è in altre parole il cuore della biochimica.
Per fare un esempio concreto e molto attuale in questi giorni, i vaccini contro la Covid-19 si basano proprio sulla proteina Spike che è presente sulla superficie del virus SARS-CoV-2 dandogli quell’effetto “corona”, la cui struttura è stata individuata in questi mesi con i metodi tradizionali (nonostante anche lì DeepMind abbia cercato di dare una mano con i suoi modelli AI). È proprio scoprendo la struttura della proteina che si è stati in grado di realizzare i vaccini.
Ma non solo, si pensa che un errato ripiegamento proteico sia alla base di svariate patologie come il morbo di Alzheimer e il diabete mellito di tipo 2, il morbo di Parkinson o la fibrosi cistica. Conoscere la struttura dei cristalli proteici potrebbe essere la chiave per creare delle cure mirate.
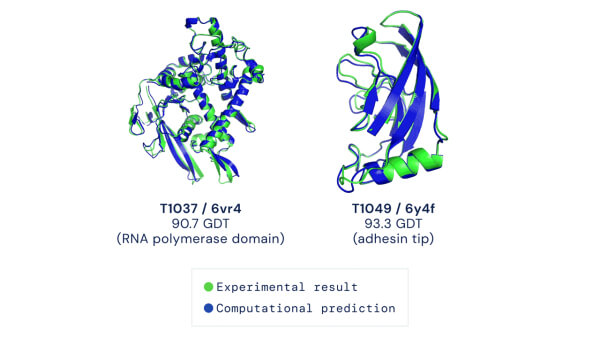
Due esempi di target proteici nella categoria della modellazione libera. AlphaFold prevede strutture altamente accurate rispetto al risultato sperimentale. Fonte: DeepMind
Ma quali sono i metodi tradizionali usati finora, e perché si sentiva il bisogno di un miglioramento?
Attualmente per studiare la struttura di una proteina si usano principalmente tre tecnologie, la cristallografia a raggi X (usata fin dagli anni Cinquanta), la microscopia crioelettronica e la spettroscopia a risonanza magnetica nucleare. Sono tutte tecniche valide (la proteina Spike del SARS-CoV-2 è stata trovata con la microscopia crioelettronica) ma hanno due grandi difetti: sono lente e molto costose. Tanto lente e costose da lasciarci ancora oggi senza informazioni sulla struttura della maggior parte delle proteine esistenti. Vi sono poi diverse proteine, come le proteine di membrana, difficili da cristallizzare e quindi da determinare in maniera sperimentale.
Il CASP, cos’è, cosa fa
Per tutte queste ragioni nel 1994 il biologo computazionale John Moult e altri colleghi crearono il CASP, Critical Assessment of protein Structure Prediction, una serie di esperimenti (ma sarebbe forse più corretto chiamarle competizioni) dove varie squadre fanno a gara per individuare, per mezzo di software creati ad-hoc, il ripiegamento di una serie proteine la cui struttura è già nota ma non pubblica. Il software che più si avvicina alle reali strutture proteiche vince la competizione. Scopo degli organizzatori ovviamente è stimolare la ricerca per questo problema e trovare sistemi software che riescano a sostituire i metodi utilizzati finora.
Il CASP si tiene ogni due anni dal 1994 (con un’edizione speciale aggiuntiva tenutasi quest’anno per analizzare il SARS-CoV-2) e nel 2018, al CASP 13, DeepMind presentò il suo sistema AlphaFold vincendo la competizione. Nonostante la vittoria però AlphaFold non arrivò neanche vicino all’obiettivo di 90 GDT_TS di media, considerato il traguardo oltre il quale il risultato del software è equiparabile a quello dei metodi tradizionali. La prima versione del software di DeepMind (che nel frattempo ha reso pubblico il codice su GitHub) raggiunse un GDT_TS mediano di 68,5, abbastanza per assicurarsi il primo posto ma molto lontano da quel traguardo di 90 che renderebbe il modello interessante per biologi e ricercatori che studiano il ripiegamento delle proteine.
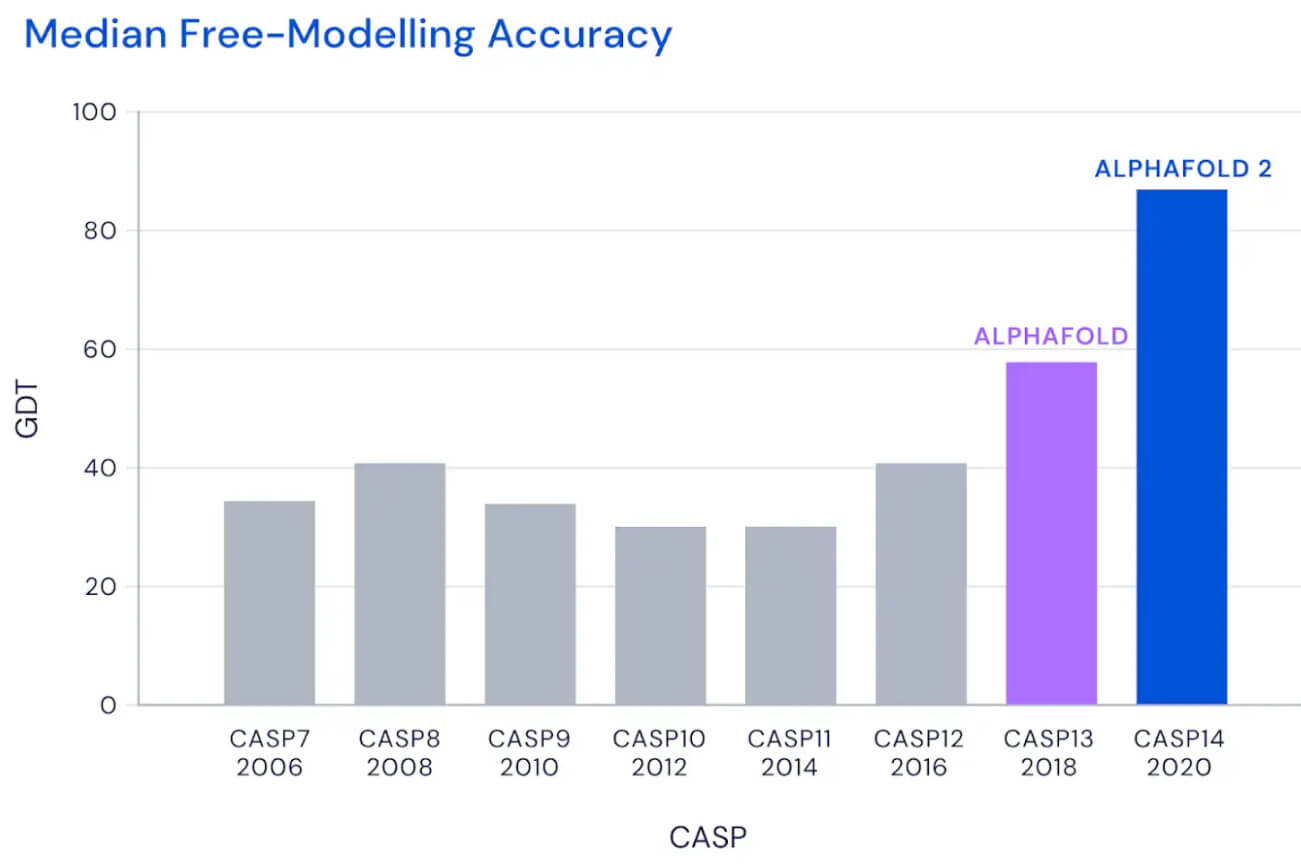
Punteggio del miglior team nella categoria “free modelling” in ogni CASP. Nel 2018 AlphaFold ottenne in questa categoria un punteggio di 58,9. Nel 2020 ha superato i 90 punti. Fonte: DeepMind
AlphaFold, prevedere la struttura delle proteine
Quest’anno tuttavia, al CASP 14, DeepMind ha presentato un software completamente riprogettato. AlphaFold nel 2018 era essenzialmente un insieme di modelli abbastanza standard (il principale era un classificatore di immagini), come spiega il capo del progetto John Jumper in un articolo di Fortune: un modello prevedeva la distanza tra le coppie di amminoacidi, un altro modello cercava di determinare gli angoli più probabili, infine il terzo migliorava la struttura complessiva.
AlphaFold invece è stato progettato da zero e con l’obiettivo specifico di prevedere la struttura delle proteine. Il sistema non si avvale più di tre distinte reti neurali: esso prende l’informazione genetica come input e rilascia la struttura 3D della proteina come output, ottenendo nelle prove del CASP 14 un punteggio mediano di 92,4 GDT, che sorpassa il limite di 90 posto dallo stesso Moult e consente al software di affermarsi come una reale alternativa ai metodi tradizionali. Con il vantaggio ovviamente che per prevedere il ripiegamento proteico AlphaFold2 richiede pochi giorni (a volte poche ore) e molte meno risorse, confrontato con i mesi o anni dell’analisi sperimentale.
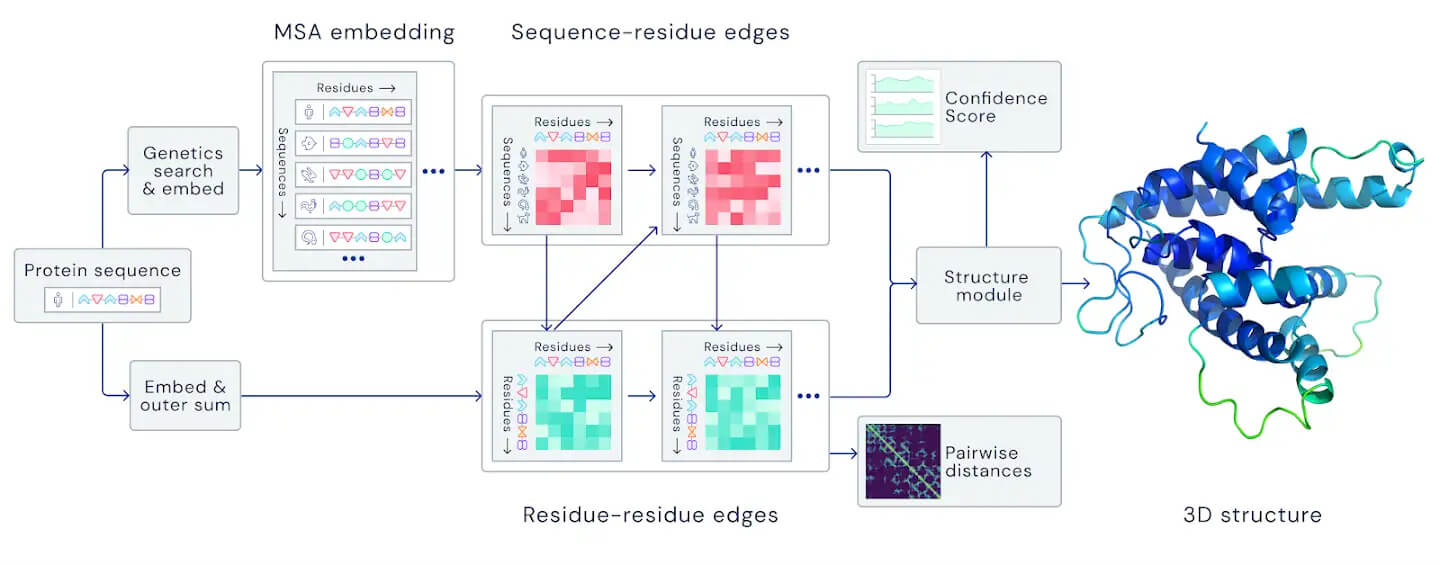
Panoramica della principale architettura della rete neurale. Il modello opera su sequenze proteiche evolutivamente correlate così come coppie di residui di amminoacidi, passando iterativamente informazioni tra le due rappresentazioni per generare una struttura. Fonte: DeepMind
DeepMind ha addestrato il sistema su dati pubblici di circa 170.000 strutture proteiche, insieme a grandi database contenenti sequenze proteiche di struttura sconosciuta. Nel training AlphaFold2 ha utilizzato circa 16 TPUv3 (ovvero 128 core TPUv3, approssimativamente equivalenti a circa 100 o 200 GPU) e la durata dell’addestramento è stata di poche settimane.
AlphaFold, reazioni positive
Molte le reazioni entusiaste nel mondo della ricerca, fra cui lo stesso Moult, che ha fatto notare come dopo il buon risultato di AlphaFold nel 2018 quest’anno almeno metà dei team partecipanti hanno iniziato a usare qualche forma di deep learning. La maggior parte delle squadre provengono dal mondo accademico, ma anche aziende tecnologiche come Tencent e Microsoft hanno deciso di partecipare al CASP 14.
Il biologo strutturale Venki Ramakrishnan, premio Nobel per la chimica nel 2009, ha definito il sistema AI “un sorprendente progresso nel problema del ripiegamento proteico“, avvenuto con decenni di anticipo rispetto a quanto molti esperti di settore avevano previsto.
Secondo Jane Thornton, ex direttrice dell’Istituto Europeo di Bioinformatica presso il Laboratorio Europeo di Biologia Molecolare nonché esperta di strutture proteiche, sentita dal MIT Technology Review, il livello di efficacia raggiunto dal sistema di DeepMind potrebbe aprire la strada alla mappatura dell’intero proteoma umano, ovvero l’insieme di proteine all’interno del nostro corpo. Oggi solo un quarto di esse sono usate come target per i farmaci, cosa che lascia il restante 75% disponibile per nuove scoperte, creando enormi occasioni per studiare nuove medicine.
Per molti esperti ed entusiasti di intelligenza artificiale, infine, i risultati raggiunti da AlphaFold sono solo un esempio delle vaste opportunità che grazie all’AI possiamo esplorare, per cercare di migliorare le nostre vite, velocizzare il progresso scientifico e approfondire la conoscenza di noi stessi. Magari cominciando proprio dalle proteine.